Wenn es im Moment einen Wissenschaftsbereich gibt, der im Monats- oder gar Wochentakt spektakuläre neue Erkenntnisse liefert, dann ist das die Erforschung alter DNA (aDNA). Allein in den vergangenen Tagen sind wieder mehrere Studien erschienen, die neues Licht auf unsere Vergangenheit werfen.
So haben US-Forscher anhand der starken genetischen Verarmung am Y-Chromosom im Fachblatt "Nature Communications" gezeigt, dass es vor circa 7.000 Jahren rund um den Planeten einen massiven Männerschwund gegeben haben muss. Ein Mann kam damals vermutlich auf 17 Frauen – verantwortlich für das große Männersterben dürften ständige kriegerische Auseinandersetzungen zwischen Clans gewesen sein, doch das sind nur Vermutungen.
Ersten Isländer und Ursprünge des Inka-Adels
Ein Artikel im Magazin "Science" schreibt kurzerhand die Geschichte der Besiedlung Islands neu: Nach der DNA-Analyse rund 1000 Jahre alter Gebeine zeigten isländische Genetiker, dass nur die Hälfte der Ur-Isländer, die rund um das Jahr 900 die Insel erreichten, Wikinger aus Norwegen und Schweden waren, während die andere Hälfte – meist Frauen – aus Schottland und Irland kam. Und obwohl die Siedler danach weitgehend unter sich blieben, kam es zu einer rätselhaften Verstärkung des skandinavischen DNA-Anteils, der heute 70 Prozent ausmacht.
Eine Studie peruanischer Forscher konnte – ebenfalls anhand genetischer Analysen von aDNA und derjenigen von heute lebenden Menschen – die Mythen vom Ursprung der Inka-Herrscher bestätigen: Der frühe Adel der südamerikanischen Hochkultur dürfte tatsächlich aus der Gegend von Cuzco und des Titicacasees stammen.
In der DNA die Vergangenheit lesen
Dieser verblüffende Boom geht auf die Forschungen eines schwedischen Forschers zurück, dem noch als Doktorand in den 1980er-Jahren die Klonierung der DNA einer ägyptischen Mumie gelang. 1997 schaffte Svante Pääbo (siehe aktuelles STANDARD-Interview) dann die Sensation: Er konnte Teile der uralten DNA eines Neandertalers sequenzieren. Das eröffnete völlig neue Möglichkeiten, in der Vergangenheit des Menschen zu lesen.
Bild nicht mehr verfügbar.
Die genetische Rekonstruktionsarbeit, die Paläogenetiker dabei leisten müssen, ist enorm. Die Älteren unter uns, die ohne Smartphone aufgewachsen sind, können es sich in etwa so vorstellen: Man versucht, Namen und Telefonnummern aus einem mehrbändigen Telefonbuch zu rekonstruieren, das man zuvor allerdings geschreddert hat, mit Altpapierschnitzeln vermischte und auf einer Mülldeponie verrotten ließ.
Dass diese aDNA-Analysen heute nicht nur möglich, sondern fast schon zur Routine geworden sind, vergleicht Ron Pinhasi mit dem methodischen Durchbruch durch die Entdeckung der Radiokarbonmethode 1949. "Auch die C14-Datierung revolutionierte die archäologischen und paläontologischen Forschungen, weil man damit das Alter der Funde unter 30.000 Jahren erstmals genau bestimmen konnte", sagt der Forscher, der seit kurzem am Department für Anthropologie der Universität Wien forscht und an etlichen Entdeckungen der letzten Jahre beteiligt war.
Das Neandertaler-Genom und wir
Was man in den vergangenen Jahren alles mit aDNA-Analysen entdeckte, ist kaum zu überblicken: Mit der Sequenzierung des Neandertaler-Genoms, die 2009 abgeschlossen war, und Vergleichen mit der DNA heute lebender Menschen wurde etwa offensichtlich, dass die meisten Menschen außerhalb Afrikas rund zwei Prozent von Neandertaler-DNA in sich tragen, weil sich unsere Vorfahren mit Neandertalern paarten. Zudem ließ sich errechnen, dass die letzten gemeinsamen Vorfahren von uns und den Neandertalern vor rund 500.000 Jahren lebten.
Pääbo und seinem Team vom 1997 gegründeten Max-Planck-Institut für evolutionäre Anthropologie in Leipzig, dem führenden Zentrum für diese Untersuchungen, gelang 2010 eine weitere Sensation: Aus einem winzigen 40.000 Jahre alten Fingerknochen, der in einer Höhle im südsibirischen Altai-Gebirge gefunden worden war, konnten die Forscher gut erhaltene DNA gewinnen, die sich sowohl vom Neandertaler wie auch vom modernen Menschen unterschied: Der Denisova-Mensch war entdeckt worden!
Bild nicht mehr verfügbar.
Eine Menschenform fast ohne Fossilien
Obwohl man von diesem bis heute nicht mehr als drei kleine Fossilien aus der Denisova-Höhle kennt (neben dem Fingerknochen noch zwei Backenzähne), weiß man mittlerweile dank verschiedenster Genom-Vergleiche eine ganze Menge über diese mysteriöse Menschenform, die noch in der Altsteinzeit in Asien lebte: Die Denisova-Menschen paarten sich mit dem Neandertaler und dem modernen Menschen, ihre DNA macht heute noch bis zu fünf Prozent des Genoms der Melanesier aus, also der Bewohner von Papua-Neuguinea und den umliegenden Inseln.
Erst vor wenigen Monaten entdeckte man aufgrund von Genomvergleichen heute lebender Asiaten, dass es noch eine zweite, genetisch etwas andere Population von Denisova-Menschen gegeben haben muss, die vor allem Spuren in der Erbsubstanz der Chinesen und Japaner hinterlassen hat. Von dieser Denisovaner-Population gibt es noch nicht einmal ein einziges Fossil.
Doch das ist die Ausnahme, wie Ron Pinhasi erklärt: "Natürlich sind wir weiter auf alte Knochen angewiesen, um daraus aDNA-Proben zu ziehen." Das stößt aber an Grenzen, denn die Erbsubstanz zerfällt insbesondere in warmen, feuchten Gegenden schneller. Pinhasi will es trotzdem mit solchen Knochen weiter versuchen, ist er doch einer der weltweit führenden Experten in diesem Spezialbereich: So entdeckte er 2014, dass sich im sogenannten Felsenbein, dem härtesten Knochen des Menschenschädels, die DNA 100-mal besser erhält als in anderen Knochen.
Exponentiell wachsende Datenmenge
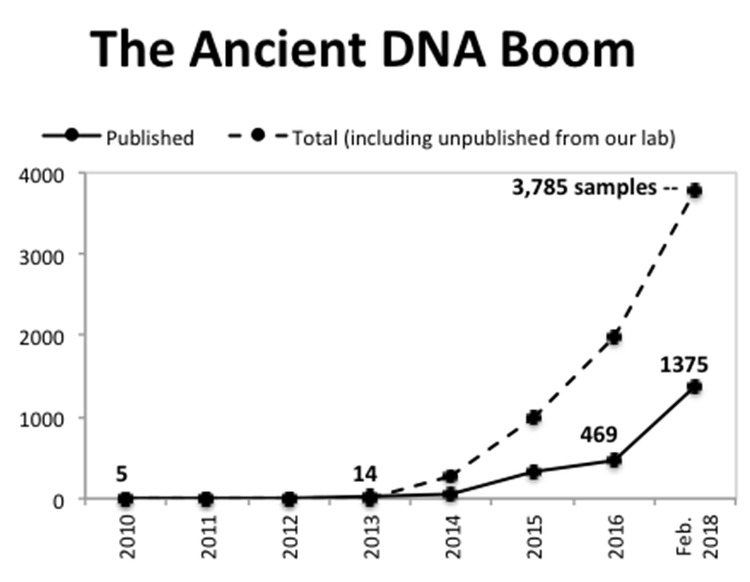
In den vergangenen Jahren nahm die Forschung dann noch einmal so richtig Fahrt auf: Lagen 2013 gerade einmal 14 aDNA-Proben unserer Ahnen vor, so waren es im Februar 2018 bereits 3785, wie David Reich in seinem kürzlich erschienenen Buch "Who We Are and How We Got Here" nicht ohne Stolz schreibt. Die meisten davon wurden nämlich im Labor des Genetikers an der Uni Harvard sequenziert.
Auch aus diesem Grund ist Reich einer der produktivsten Protagonisten dieses Forschungsbereichs und wartete mit etlichen neuen Erkenntnissen über die Migrationsbewegungen des Menschen auf. Allein in diesem Jahr konnte er mit verschiedenen Forscherteams unter anderem zeigen, dass es in Nordamerika eine bisher unbekannte Einwanderergruppe gegeben hat, dass die Besiedlung der südpazifischen Inseln vor rund 3000 Jahren in zwei Wellen erfolgte – einer aus dem heutigen Taiwan und einer aus Indonesien – und wie und wann sich die Landwirtschaft in Ostasien durch Zuwanderer ausbreitete.
An dieser zuletzt genannten Studie, die erst im Mai erschien, war auch Ron Pinhasi beteiligt, der es als großes Privileg empfindet, als Forscher "Teil dieser extrem spannenden Zeit der Entdeckungen zu sein". Er warnt aber auch davor, alles aus der alten DNA ablesen zu wollen: "Wenn es um kulturelle Aspekte der Menschheitsentwicklung geht – und fast alles hat mit Kultur zu tun -, brauchen wir zunächst einmal archäologische Informationen. Von deren Qualität hängt es ab, wie gut die Forschung in Kombination mit den DNA-Daten ist."
Rass(ist)ische Fehlinterpretationen
Gerade bei der Zusammenführung von biologischen und kulturellen Informationen kam es in jüngster Zeit zu einigen Debatten innerhalb der Community. Es ging um die Frage, ob man bestimmte Kulturen oder Populationen mit bestimmten genetischen Merkmalen korrelieren kann und soll. Und hier wird die Forschung dann auch politisch oder besser: anfällig für politisch motivierte Fehlinterpretationen im Sinne von "Rassen" und "Rassenunterschieden".
"Für mich und so gut wie alle meine Kollegen ist Rasse kein wissenschaftliches Konzept. Und Punkt", sagt Pinhasi kategorisch und verweist auf einige der grundlegenden Erkenntnisse aus der genetischen Erforschung des Menschen: dass es nämlich mehr DNA-Variationen innerhalb der verschiedenen, ohnehin nicht klar abzugrenzenden Populationen gibt als zwischen den Populationen.
Das wiederum hat damit zu tun, dass Migration und Vermischung quasi zur DNA des Menschen gehört, weshalb sich ihre vielfältigen Spuren auch in unseren Genen wiederfinden. "Menschliche Wanderungsbewegungen haben einfach ständig stattgefunden", sagt Pinhasi, für den die Flüchtlingskrise – aus längerfristiger Perspektive der Menschheitsgeschichte betrachtet – nichts Außergewöhnliches ist: "Der einzige Unterschied ist, dass heute in absoluten Zahlen mehr Menschen unterwegs sind, weil heute die Bevölkerungszahlen so viel höher sind. Nimmt man aber die relativen Anteile, dann waren früher in den meisten Epochen wahrscheinlich weit mehr Leute unterwegs als heute." (Klaus Taschwer, 8.6.2018)